Os métodos
utilizados foram:
1) mapa de concentração do teor de ''lead' na área mapeada em uma das margens do Rio Meuse;
2) mapa base;
3)
sumário da variável;
4)
histograma;
5)
box-plot;
6)
variograma experimental;
7)
variograma ajustado do obtido em 6);
8)
dispersograma das variáveis;
9)
cálculo do coeficiente de correlação;
10)
variação do teor de chumbo;
11) mapa
de isoteores (isopletas, contornos);
12) mapa
de contornos preenchidos.Resultados:
1) Mapa de concentração do teor de ''lead' na área mapeada em uma das margens do Rio Meuse:
Foi necessário estimar os parâmetros de um modelo esférico omnidirecional de semivariograma que foram utilizados no programa de krigagem. Com as simulações, obteve-se:
Comando:
s.grid<-GridTopology(c( 178500, 329965),c(60,60),c(30,30))s.grid<-SpatialPoints(s.grid)gridded(s.grid)<-TRUEdata(meuse.all)v<-vgm(0.56439420,"Sph",535.8096,0.07655493)kr<-krige(lead~1,~x+y,model=v,data=meuse.all,newd=s.grid,nsim=3)spplot(kr["sim2"],xlab="Xloc(0-4)",ylab="Yloc(0-4)",main="Simulação Gaussiana 1")
Comando:
s.grid<-GridTopology(c( 178500, 329965),c(60,60),c(30,30))s.grid<-SpatialPoints(s.grid)gridded(s.grid)<-TRUEdata(meuse.all)v<-vgm(0.56439420,"Sph",535.8096,0.07655493)kr<-krige(lead~1,~x+y,model=v,data=meuse.all,newd=s.grid,nsim=3)spplot(kr["sim1"],xlab="Xloc(0-4)",ylab="Yloc(0-4)",main="Simulação Gaussiana 2")x11()
Mapa de concentração obtido baseado em krigagem simples:
Comando:
library(gstat)data(meuse.all)coordinates(meuse.all) = ~x+ydata(meuse.grid)gridded(meuse.grid) = ~x+y# grid já veio definidom <- vgm(.59, "Sph", 874, .04)x <- krige(log(lead)~1, meuse.all, meuse.grid, model = m,beta=5.9)l2 = list("SpatialPolygonsRescale", layout.north.arrow(), offset = c(181300,329800),scale = 400)spplot(x["var1.pred"], sp.layout=list(l2), col.regions=bpy.colors(20),key.space=list(x=0.1,y=.95,corner=c(0,1)), main = "simples kriging predictions", xlab="Xloc",ylab="Yloc ")
Comando:
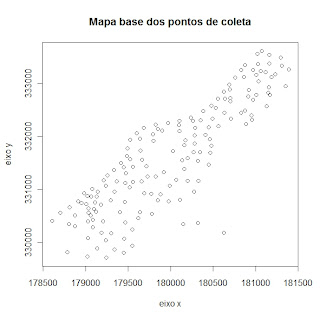
data(meuse.all)
plot(meuse.all[,2],meuse.all[,3],xlab='eixo x',ylab='eixo y',main='Mapa base dos pontos de coleta')
3) Sumário da variável:
4) Histograma:
Comando:
Comando:
library(gstat)
data(meuse.all)
attach(meuse.all)
lead
pb=lead
summary(pb)
Min. 1st Qu. Median Mean 3rd Qu. Max.
27.00 68.75 116.00 148.60 201.80 654.00
Comando:
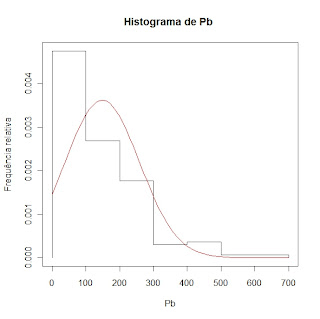
data(meuse.all)
g <- meuse.all
x.norm<- g$lead
h<-hist(x.norm,breaks=8)
xhist<-c(min(h$breaks),h$breaks)
yhist<-c(0,h$density,0)
xfit<-seq(0, 700, by=10)
yfit<-dnorm(xfit,mean=mean(x.norm),sd=sd(x.norm))
plot(xhist,yhist,type="s",ylim=c(0,max(yhist,yfit)),xlab="Pb",ylab="Frequência relativa",main="Histograma de Pb ")
lines(xfit,yfit,col="red")
5) Box-plot:
Comando:
Comando:
data(meuse.all)
vgm1<-variogram(log(lead)~1, locations=~x+y, data=meuse.all)
x=range(vgm1[,2])
y=range(vgm1[,3])
plot(x,y, asp = 1000, type = "n", main="Ajuste de um modelo teórico ao semivariograma")
points(vgm1[,2],vgm1[,3],col="blue",cex=1.5)
lines(vgm1[,2],vgm1[,3],col="blue")
f<-fit.variogram(vgm1,vgm(0.16, "Exp", 1500, 0.5))
v<-vgm(f$psill[2], "Exp",f$range[2],f$psill[1])
ff<-variogramLine(v,maxdist=1543 ,n = 15 , min = 80)
points(ff[,1],ff[,2],col = "red")
lines(ff[,1],ff[,2], col = "red")
asp=max(x)max(y)
asp=1542.27/0.6211689=2482.851
6) Variograma experimental:
Comando:
Comando:
data(meuse.all)
g=gstat(id='lead',formula=log(lead)~1,locations=~x+y,data=meuse.all)
grafico=variogram(g)
plot(grafico,main=' Variograma experimental de Pb',xlab='Distância',ylab='Semivariância')
7) Variograma ajustado do obtido em 6):
Comando:
Comando:
data(meuse.all)
vgm1<-variogram(log(lead)~1, locations=~x+y, data=meuse.all)
x=range(vgm1[,2])
y=range(vgm1[,3])
plot(x,y, asp = 1000, type = "n", main="Ajuste de um modelo teórico ao variograma")
points(vgm1[,2],vgm1[,3],col="blue",cex=1.5)
lines(vgm1[,2],vgm1[,3],col="blue")
f<-fit.variogram(vgm1,vgm(0.16, "Exp", 1500, 0.5))
v<-vgm(f$psill[2], "Exp",f$range[2],f$psill[1])
ff<-variogram.line(v,maxdist=1543 ,n = 15 , min = 80)
points(ff[,1],ff[,2],col = "red")
lines(ff[,1],ff[,2], col = "red")
model psill range
1 Nug 0.07655493 0.0000
2 Exp 0.56439420 535.8096
asp=max(x)max(y)
asp=1542.27/0.6211689=2482.851
asp=max(x)max(y)
asp=1542.27/0.6211689=2482.851
Adotamos o valor 1000 por se tratar de ordem de grandeza
8) Dispersograma das variáveis:
9) Cálculo do coeficiente de correlação:
10) Variação do teor de chumbo:
Com os mapas obtidos podemos observar que as concentrações do teor de chumbo estavam maiores quanto mais próximo ao leito do rio.
Com os mapas obtidos podemos observar que as concentrações do teor de chumbo estavam maiores quanto mais próximo ao leito do rio.
11) Mapa de isoteores:
Comando:
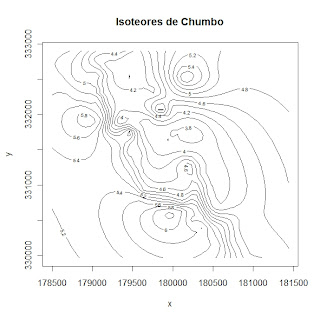
12) Mapa de contornos preenchidos:Comando:
s.grid<-GridTopology(c( 178500, 329965),c(60,60),c(50,50))
s.grid<-SpatialPoints(s.grid)
#spatial points
data(meuse.all)
m <- vgm(0.5643942, "Exp", 535.8096, 0.07655493)
xx <- krige(log(lead)~1, ~x+y, model = m, data = meuse.all, newd = s.grid)
dfxx<-as.data.frame(xx)
mz<-matrix(dfxx[,3], nrow=50, ncol=50, byrow=FALSE)
X11()
contour(x =seq(178500,181440,by= 60), y=seq(329965,332905,by=60),mz,nlevels=10,
xlab="x",ylab="y",main="isoteores de Chumbo") 
Comando:
s.grid<-GridTopology(c(178500, 329965),c(60,60),c(50,50))
s.grid <- SpatialPoints(s.grid)
data(meuse.all)
m <- vgm(0.5643942, "Exp", 535.8096, 0.07655493)
xx <- krige(lead~1, ~x+y, model = m, data = meuse.all, newd = s.grid)
dfxx <- as.data.frame(xx)
mz <- matrix(dfxx[,3], nrow=50, ncol=50, byrow=FALSE)
nmz <- matrix(nrow=50, ncol=50)
for (i in 1:50)
for (j in 1:50)
{nmz[i,j]=mz[i,51-j]}
x =seq(1.0,5.9,by= 0.1)
y=seq(1,5.9,by=0.1)
filled.contour(x, y,nmz,nlevels=10,color=terrain.colors, xlab="X",ylab="Y",main="Mapa de Isoteores de Chumbo")







Nenhum comentário:
Postar um comentário